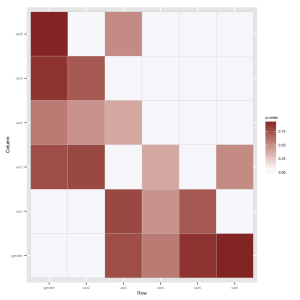
… ridiculously photogenic factors (heatmap with p-values)
Want to share your content on R-bloggers? click here if you have a blog, or here if you don't.
Some months ago, I had to explore a vast amount of categorical variables before making some multivariate analyses.
One good way to know your raw data, to make new hypotheses…etc, is to calculate some pairwise “crude” chi-square tests of independence of your factors, but it can be very time-consuming.
I mean, not time-consuming to make the tests (with a simple command it can be done), but to revise all of them. My memory (not R’s) failed after 10 tests, and I had 22 factors…
Now that I have a bit more experience in R cooking, I tried to put in practice one idea: what if I could look at all of these p-values at a glance? Inmediately thought of a plot.
What kind…of…plot? Inmediately thought of HEATMAPS. Why? If I managed to get my p-values (alpha errors) in a matrix, a heatmap could help me to rapidly identify significant ones, or even make clusters of significant associations. Although it cannot lead to formal conclusions, a heatmap could be very helpful.
Here is how I could print a heatmap made of p-values. First of all, let’s construct some data (post inspired in this one):
library(plyr) library(ggplot2) library(scales) library(reshape)
gender <- c( "1", "2", "1", "2", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "2", "2", "1", "1", "1", "2", "1", "2", "1", "1", "1", "1", "1", "1", "1", "2", "2", "1", "2", "1", "1", "1", "1", "1", "1", "2", "1", "1", "1", "1", "1", "1", "2", "2", "1", "2", "1", "2", "2", "1", "1", "1", "2", "1", "2", "2", "2", "2", "2", "1", "1", "1", "1", "1", "1", "1", "1", "2", "1", "1", "1", "2", "1", "1", "1", "1", "2", "1", "1", "1", "1", "1", "2", "1", "2", "1", "1", "1", "1", "1", "1", "1", "2", "1", "1", "1", "2", "1", "1", "2", "1", "1", "2", "2", "2", "1", "2", "1", "2", "1", "1", "1", "1", "2", "2", "2", "2", "2", "1", "1", "1", "1", "2", "2", "1", "1", "1", "1", "1", "1", "2", "1", "1", "2", "1", "2", "1", "1", "1", "1", "1", "2", "1", "1", "1", "1", "1", "2", "1", "1", "2", "1", "2", "1", "1", "1", "1", "1", "2", "1", "2", "2", "1", "2", "2", "1", "1", "1", "1", "1", "2", "2", "1", "1", "1", "2", "1", "2", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "2", "1", "1", "1", "2", "2", "1", "2", "1", "1", "1", "1", "2", "1", "1", "1", "1", "1", "1", "2", "2", "1", "1", "1", "1", "2", "1", "1", "1", "1", "2", "1", "2", "1", "1", "2", "1", "1", "1", "1", "1", "2", "2", "1", "1", "1", "1", "1", "1", "1", "1", "1", "2", "1", "2", "1", "1", "1", "2", "1", "2", "2", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "2", "1", "2", "1", "1", "1", "1", "2", "1", "1", "1", "2", "1", "2", "2", "1", "1", "1", "1", "2", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "2", "1", "1", "1", "2", "1", "1", "1", "2", "2", "1", "1", "2", "2", "1", "1", "1", "2", "1", "1", "1", "1", "1", "2", "1", "1", "1", "1", "1", "2", "1", "2", "2", "1", "1", "1", "1", "1", "2", "1", "2", "1", "1", "1", "1", "1", "1", "2", "1", "2", "1", "1", "1", "1", "1", "1", "2", "2", "1", "1", "2", "1", "2", "2", "1", "2", "2", "1", "2", "2", "2", "1", "2", "1", "2", "2", "1", "1", "1", "1", "1", "1", "1", "1", "1", "2", "2", "1", "1", "2", "2", "2", "1", "2", "2", "2", "2", "1", "2", "1", "1", "2") var2 <- c( "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "0", "0", "1", "1", "0", "0", "0", "0", "1", "0", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "1", "1", "0", "0", "0", "0", "1", "1", "1", "1", "0", "0", "1", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "0", "0", "0", "0", "1", "0", "1", "0", "0", "1", "1", "1", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "1", "1", "1", "0", "0", "1", "1", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "0", "1", "0", "1", "1", "0", "0", "0", "0", "0", "1", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "1", "1", "1", "0", "0", "1", "0", "1", "0", "1", "0", "0", "1", "0", "0", "0", "1", "0", "0", "1", "1", "0", "0", "0", "0", "1", "0", "1", "1", "0", "1", "1", "1", "0", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "1", "0", "1", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "0", "0", "1", "1", "0", "1", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0") var3 <- c( "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "1", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "0", "0", "0", "1", "1", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "1", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "1", "0", "0", "0", "1", "1", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "1", "1", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "1", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "0", "1", "0", "1", "0", "1", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "1", "0", "1", "0", "1", "1", "1", "1", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "0", "0", "0", "1", "0", "0", "0", "1", "0") var4 <- c( "0", "1", "0", "0", "0", "0", "1", "0", "1", "1", "0", "1", "0", "1", "1", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "1", "1", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "1", "1", "0", "1", "0", "1", "1", "1", "0", "1", "0", "0", "0", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "1", "1", "1", "1", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "1", "1", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "1", "0", "0", "0", "1", "0", "1", "1", "1", "1", "0", "1", "1", "1", "1", "1", "1", "1", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "1", "1", "1", "1", "1", "1", "0", "0", "0", "1", "0", "1", "0", "0", "0", "1", "1", "0", "0", "1", "0", "1", "0", "1", "0", "0", "1", "1", "1", "1", "1", "0", "0", "0", "0", "0", "0", "1", "0", "1", "1", "0", "1", "1", "0", "1", "0", "1", "0", "1", "0", "0", "0", "0", "1", "1", "1", "1", "0", "0", "1", "1", "1", "1", "1", "0", "1", "0", "1", "1", "1", "1", "0", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "1", "1", "1", "1", "1", "0", "1", "1", "1", "0", "1", "0", "0", "1", "1", "1", "1", "0", "0", "1", "1", "0", "0", "1", "1", "0", "0", "1", "0", "1", "1", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "0", "0", "1", "1", "0", "0", "0", "1", "0", "1", "0", "0", "0", "1", "0", "0", "1", "1", "1", "1", "1", "0", "1", "1", "1", "0", "1", "1", "1", "0", "0", "1", "1", "1", "1", "1", "0", "1", "1", "1", "0", "1", "1", "0", "0", "0", "0", "0", "1", "0", "1", "0", "1", "1", "1", "1", "0", "1", "1", "0", "1", "0", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "0", "0", "1", "1", "1", "0", "0", "0", "0", "1", "1", "1", "1", "1", "1", "1", "1", "0", "1", "1", "1", "1", "0", "1", "0") var5 <- c( "0", "1", "1", "0", "0", "1", "0", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "0", "1", "0", "0", "1", "0", "0", "1", "1", "0", "0", "0", "0", "0", "1", "0", "0", "1", "0", "1", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "1", "1", "0", "1", "1", "0", "1", "1", "1", "1", "1", "1", "0", "1", "1", "0", "0", "1", "1", "0", "1", "0", "0", "0", "1", "0", "1", "1", "1", "0", "0", "0", "1", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "1", "0", "1", "0", "0", "1", "1", "0", "0", "1", "1", "1", "0", "0", "0", "0", "0", "0", "1", "1", "0", "1", "1", "1", "1", "0", "1", "0", "1", "1", "0", "0", "0", "0", "1", "1", "1", "1", "0", "1", "1", "0", "0", "1", "0", "1", "1", "0", "1", "1", "0", "1", "0", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "1", "1", "1", "0", "1", "0", "0", "1", "0", "1", "0", "0", "0", "1", "1", "0", "0", "1", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "0", "0", "1", "0", "0", "0", "0", "0", "1", "0", "1", "0", "0", "0", "1", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "0", "1", "1", "1", "1", "0", "1", "1", "0", "1", "0", "0", "1", "1", "1", "1", "1", "1", "0", "1", "1", "0", "1", "1", "1", "0", "0", "0", "0", "0", "1", "1", "1", "0", "1", "1", "0", "0", "0", "1", "0", "0", "1", "0", "0", "0", "1", "1", "1", "1", "1", "1", "1", "0", "1", "0", "1", "1", "0", "1", "1", "0", "1", "1", "1", "0", "0", "0", "0", "1", "1", "1", "1", "0", "1", "1", "1", "0", "1", "1", "1", "1", "1", "1", "0") var6 <- c( "1", "1", "0", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "0", "0", "1", "1", "0", "0", "0", "0", "0", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "1", "1", "1", "0", "0", "0", "0", "0", "0", "1", "0", "0", "0", "1", "0", "0", "1", "1", "1", "0", "0", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "0", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "0", "1", "1", "1", "1", "0", "0", "1", "1", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0", "0", "0", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "1", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "0", "1", "1", "0", "0", "1", "1", "1", "1", "0", "0", "0", "0", "1", "0", "0", "0", "1", "1", "1", "1", "1", "1", "0", "1", "0", "0", "0", "1", "1", "0", "0", "1", "1", "0", "1", "1", "1", "1", "1", "1", "1", "1", "0", "0", "0", "1", "1", "1", "0", "1", "1", "1", "1", "0", "1", "0", "0", "1", "1", "1", "1", "0", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "1", "0") corfac <- data.frame(gender,var2,var3,var4,var5,var6) summary(corfac) class(corfac)
Now, we have our little database, in a data frame. However, for constructing a heatplot data needs to undergo some transformations:
Template of a half (triangular) matrix -> matrix with NAs:
combos <- combn(ncol(corfac),2) # combinations without repetitions
Template of a full (square) matrix -> recommended option:
combos <- expand.grid(rep(list(1:ncol(corfac)), 2 )) # combinations with repetitions combos <- as.matrix(combos) combos <- t(combos) # transpose matrix
Once made a template, this code will compute p-values pairwise, following the pattern in combos, until the matrix is filled:
mat1 <- adply(combos, 2, function(x) {
test <- chisq.test(corfac[, x[1]], corfac[, x[2]])
out <- data.frame("Row" = colnames(corfac)[x[1]]
, "Column" = colnames(corfac[x[2]])
, "Chi.Square" = round(test$statistic,3)
, "df"= test$parameter
, "p.value" = round(test$p.value, 3)
)
return(out)
})
And now, some fun! here’s the first heatmap you can make with this data:
ggplot(mat1, aes(Row, Column, fill = p.value)) +
geom_tile(colour="gray80") +
theme_gray(8) +
scale_fill_gradient2(low = muted("blue"), mid = "white", high = muted("red"),
midpoint = 0.04, space = "Lab", na.value = "grey50", guide = "colourbar")
If we want to go further, and do some clustering on your heatmap, you have to continue modifying the database (only possible with the “square” matrix, not with the “triangular”):
CLUSTERED HEATPLOT
First of all, let’s use the great reshape package, to do the following:
Select he columns of interest
matz <- mat1[,c(2,3,6)] head(matz)
Cast the matrix to have variable names as row names and column names
mat2df <- cast(matz, Row~Column) # Eureka! mat2 <- as.matrix(mat2df) head(mat2)
The matrix is ready!
Now, choose a package you like to plot your heatmap in:
My favourite, using heatmap.2:
library(gplots)
# Defining breaks for the color scale
myCol <- c("yellow", "orange", "red", "gray20", "gray15")
myBreaks <- c(0, 0.001, 0.01, 0.05, 0.8, 1)
hm <- heatmap.2(mat2, scale="none", Rowv=T, Colv=T,
col = myCol, ## using your colors
breaks = myBreaks, ## using your breaks
# dendrogram = "none", ## to suppress warnings
margins=c(5,5), cexRow=0.7, cexCol=0.7, key=FALSE, keysize=1.5,
trace="none")
legend("topleft", fill = myCol, cex=0.9,
legend = c("0 to 0.001", "0.001 to 0.01", "0.01 to 0.05", "0.05 to 0.8", ">0.8"))
Others: Heatplus
# source("http://bioconductor.org/biocLite.R") # select mirror to install
# biocLite("Heatplus") # install Heatplus
library(Heatplus)
reg1 <- regHeatmap(mat2)
plot(reg1)
Using pheatmap:
library(pheatmap) pheatmap(mat2)
Any suggestions/ tricks, etc, about color picking, or using other type of data for the heatmap (rather than p-values)…? (Some tips using residuals from here, and here some enhancements to correlation heatmaps).
R-bloggers.com offers daily e-mail updates about R news and tutorials about learning R and many other topics. Click here if you're looking to post or find an R/data-science job.
Want to share your content on R-bloggers? click here if you have a blog, or here if you don't.